In een recente studie gepubliceerd in bioRxiv* Preprint-server hebben de onderzoekers verschillende monoklonale antilichamen (mAbs) gescreend om het optimale mAb te selecteren met een hogere bindingsaffiniteit voor het receptorbindende domein (RBD) van het ernstige acute respiratoire syndroom coronavirus 2 (SARS-CoV-2) eiwitten uit verschillende varianten met behulp van in silico nadert.
Stadion: Naar een ideaal monoklonaal antilichaam met hogere bindingsaffiniteit voor het receptorbindende domein van circulerende SARS-CoV-2-eiwitten van verschillende varianten. Afbeelding tegoed: Kateryna Kon / Shutterstock.com
achtergrond
De aanhoudende pandemie veroorzaakt door SARS-CoV-2 heeft wereldwijd meer dan 311 miljoen mensen besmet en tot dusver meer dan 5,5 miljoen doden veroorzaakt. Hoewel de snelle ontwikkeling en snelle verspreiding van coronavirusziekte 2019 (COVID-19)-vaccins de ernstige SARS-CoV-2-resultaten effectief hebben verminderd, zijn grote aantallen mensen nog niet gevaccineerd vanwege ongelijke toegang tot het vaccin in delen van de wereld.
Daarom is nieuw onderzoek naar genomica, moleculaire structuur en dynamiek, associatiemechanismen en levenscyclus van SARS-CoV-2-infectie nodig om effectieve strategieën te vinden voor de behandeling van COVID-19.
over studeren
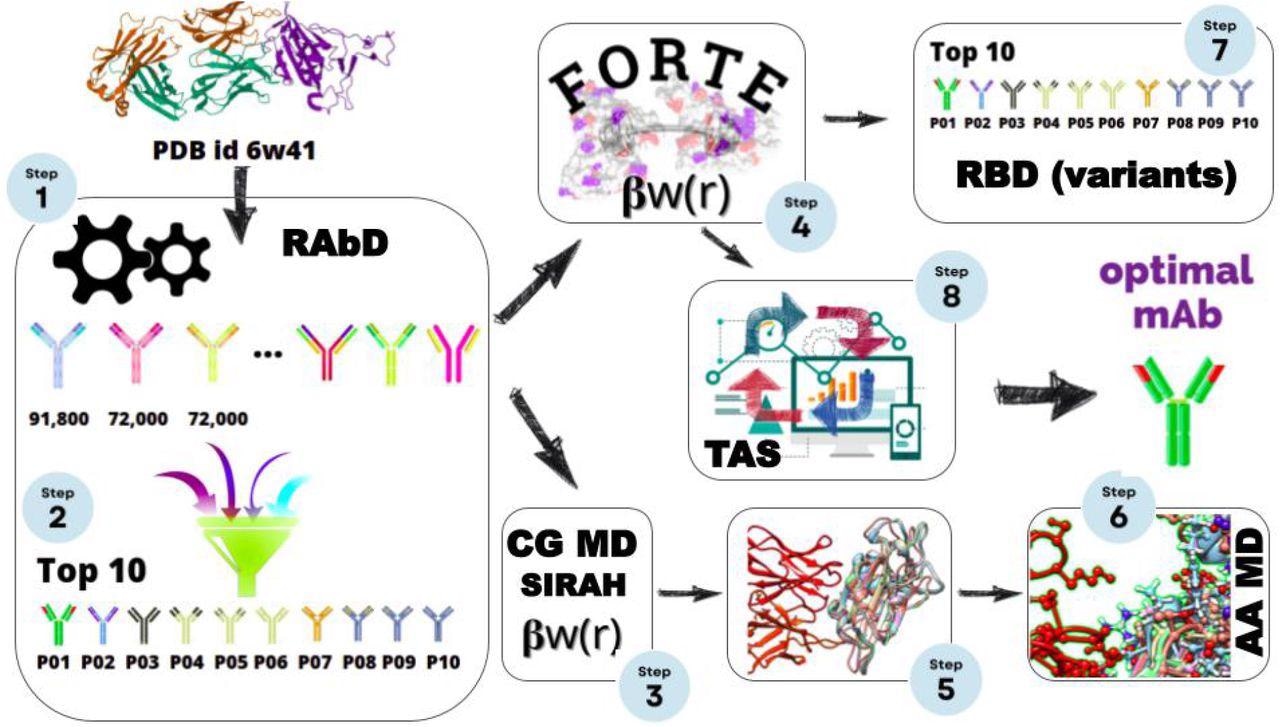
In deze studie evalueerden onderzoekers verschillende complexe antigeen-antilichaam (Ag-Ab) interacties met behulp van antilichaamontwerpsoftware, gevolgd door pH-constante Monte Carlo (MC)-berekeningen en grove-vaste lading (CG) moleculaire dynamica-berekeningen. , en een atomaire MD-simulatie met een vaste lading. Deze onderzoeken werden uitgevoerd in een poging om een Ab-kandidaat te identificeren met een relatief hogere affiniteit in alle vier soorten beoordelingen met behulp van het huidige experimentele RBD-CR3022-complex als sjabloon.
De initiële test werd uitgevoerd met behulp van de antilichaamstructuursoftware ontworpen door ROSETTA. Volgens de scorefunctie classificeerden de onderzoekers 235.000 kandidaten, waarvan er slechts 383 verbeterde affiniteiten vertoonden in vergelijking met het CR3022-RBD-complex.
Onder de 383 kandidaten selecteerden de onderzoekers tien Ag-Ab-complexen die de beste affiniteit vertoonden voor alle theoretische berekeningen. De tien geselecteerde kandidaten werden verder geëvalueerd door middel van vrije energie (G) berekeningen met behulp van universele bemonstering (VS) en FORTE.
Resultaten
Berekeningen van de vrije energie van het RBD-Ab-complex voor tien kandidaten geselecteerd met behulp van de canopy-bemonsteringsmethode toonden aan dat P01, P05, P06, P09 en P10 de bindingsaffiniteit aanzienlijk verbeterden in vergelijking met CR3022. De vrije-energiewaarde was -18,3 kcal/mol voor P01 en -7,1 kcal/mol tot -10,8 kcal/mol voor de P01-P05-P06-P09-P10-groep ten opzichte van het wildtype CR3022-antilichaam. P01 vertoonde de beste en verbeterde bindingsaffiniteit in vergelijking met de originele CR3022 en de combinatie van P05, P06, P09 en P10.
Er werd waargenomen dat de berekeningen van de vrije bindingsenergie van RBD-Ab door FORTE voor tien geselecteerde kandidaten in de volgorde P01/P06>P02/P05/P08/P09/P10>P04>P03>P05 waren. Dit gaf P01 en P06 aan als de beste kandidaten met vrije energiewaarden van respectievelijk -0,788 kb en -0,790 kb.
Schema van multiscale in silico-protocol, bestaande uit een elementaire methodologie op basis van structurele bio-informatica om macromoleculen als potentiële kandidaten te onderzoeken (stappen 1 en 2), statisch geladen CG MD (stappen 3 en 5), pH-constante CG-simulatie MC (stappen 4 en 7 en 8) en een statische lading MD-simulatie. Aan het einde van deze cyclus worden verbeterde mAbs met hogere bindingsaffiniteit verkregen.
De onderzoekers breidden de analyse uit en testten de bindingsaffiniteiten van deze kandidaten met door ROSETTA ontworpen fragmenten van mAbs met verschillende SARS-CoV-2-varianten. De resultaten onthulden een verbeterde bindingsaffiniteit voor alle 10 kandidaten in vergelijking met de oorspronkelijke CR3022. Dit geeft aan dat alle moleculen van P01 tot P10 de interactie tussen SARS-CoV-2 en de gastheercel kunnen remmen door te voorkomen dat RBD beschikbaar is om angiotensine-converterend enzym 2 (ACE2) te binden.
Interessant is dat zowel natieve CR3022 als alle door ROSETTA ontworpen bindmiddelen een hogere RBD-affiniteit vertoonden voor de SARS-CoV-2 Omicron-variant, wat suggereert dat CR3022 en zijn afgeleide mAbs de Omicron-variant kunnen neutraliseren.
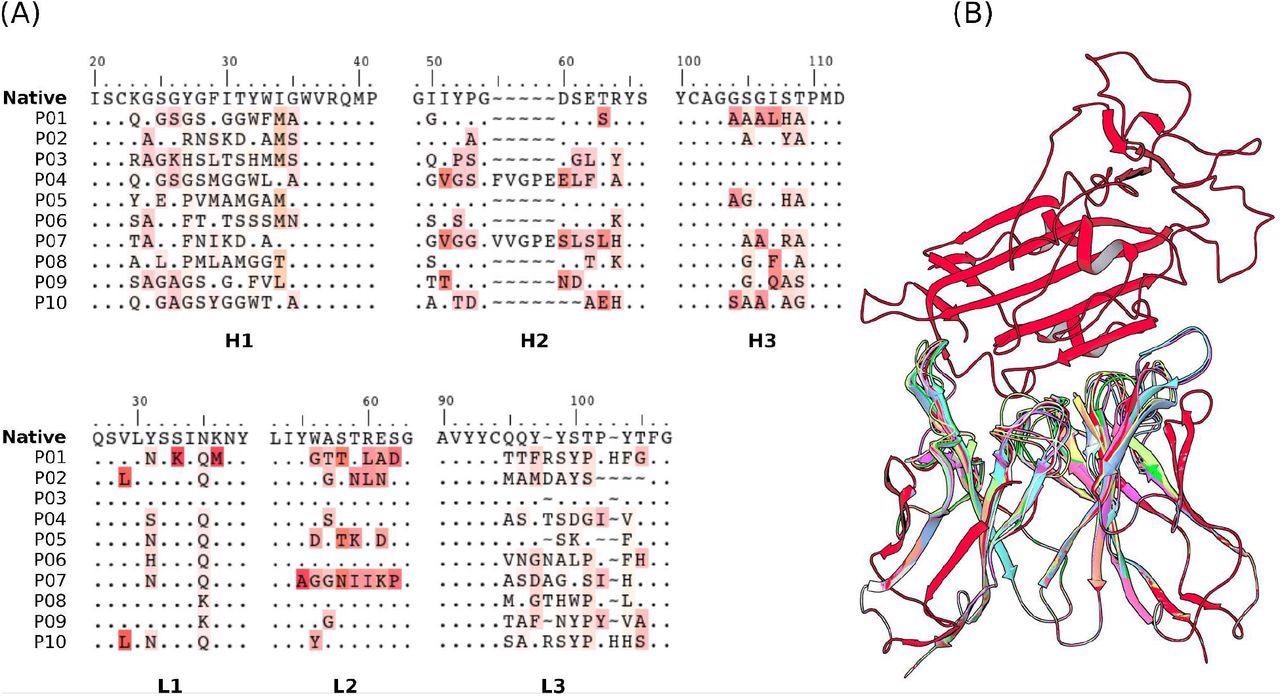
Sequenties van CDR’s (A) en structuren (B) van 10 mAbs geïdentificeerd voor beoordelingen met behulp van US/SIRAH- en FORTE-methoden. Stippen vertegenwoordigen identiteiten. De overige tekens in (A) werden gekleurd op basis van gelijkenis met hun tegenhangers in de oorspronkelijke CR3022-referentiereeks. In (B) werd RBD (boven) rood gekleurd en elk kandidaat-antilichaam (onder) een andere kleur.
De onderzoekers evalueerden verder de bijdrage van een specifiek aminozuur aan RBD-binding aan de bovenste twee binders, P01 en P06. Dit werd bereikt door het gebruik van isotopische alanine-scanning (TAS), waarbij een aminozuur van de potentiële kandidaat werd vervangen door alanine en vervolgens werd de complexe simulatie herhaald voor dit nieuwe potentiële bindmiddel.
De resultaten van dit experiment toonden aan dat aminozuren die zich op het Ag-Ab-grensvlak en diep in de eiwitstructuur bevinden, essentieel zijn voor het complexvormingsproces. Dit komt door de verstrekkende aard van elektrostatische interacties en elektrostatische koppeling tussen getitreerde samenstellingen, die worden beschreven als ‘elektrostatische epitopen’.
De onderzoekers merken ook op dat de totale nettolading van mAbs de bindingsaffiniteit kan beïnvloeden. Hiertoe vonden ze een duidelijke lineaire neiging voor mAbs met een lagere lading om een hogere bindingsaffiniteit voor RBD te vertonen. De binding van SARS-CoV-2-antigenen met de bestudeerde mAb’s werd afgeleid door de coulombkracht, die ook in eerdere onderzoeken werd waargenomen.
conclusies
Over het algemeen is de multischaalbenadering die in het huidige onderzoek wordt gebruikt, een snel, robuust en betrouwbaar hulpmiddel voor het ontwerpen van betere moleculaire liganden om het beste kandidaat-mAb voor SARS-CoV-2-varianten te identificeren, inclusief de Omicron-variant. Dit is multiband in silico Daarom is de aanpak een redelijk en realistisch hulpmiddel voor de diagnose, behandeling en preventie van SARS-CoV-2.
*Belangrijke notitie
bioRxiv Het publiceert voorlopige wetenschappelijke rapporten die niet door vakgenoten zijn beoordeeld en daarom niet als afdoend mogen worden beschouwd, die de klinische praktijk/gezondheidsgerelateerd gedrag niet mogen leiden, of als gevestigde informatie moeten worden behandeld.

“Gamedokter. Een zombiefanaat. Muziekstudio. Ninjacafé. Televisieliefhebber. Aardige fanatieke alcoholist.






More Stories
China is van plan het Tiangong-ruimtestation uit te breiden; Stel deze in op “Space Rule” omdat het ISS wordt uitgeschakeld
De Verenigde Staten detecteren het eerste geval van de H5N1-vogelgriep bij een varken, wat aanleiding geeft tot bezorgdheid voor de mens
NASA zal in 2025 de ruimtewandelingen aan boord van het internationale ruimtestation hervatten na een lek in het ruimtepak